paru dans Angew. Chem. Int. Ed., 2022
https://onlinelibrary.wiley.com/doi/10.1002/anie.202203066
Les complexes métalliques sont de plus en plus utilisés pour des applications biologiques et médicinales, notamment en tant que médicaments (métallomédicaments). Les environnements biologiques sont riches en bases de Lewis et contiennent une variété d’ions métalliques divalents qui constituent un réservoir échangeable. Les complexes métalliques exogènes peuvent donc y subir des échanges de cation métallique ou de ligand. De tels échanges diminuent leur concentration intracellulaire et/ou peuvent changer la nature de l’espèce active. Connaître la nature (ou spéciation) en contexte cellulaire est donc clef pour comprendre et améliorer l’activité biologique observée.
Les complexes de Mn(II) sont particulièrement difficiles à détecter et à quantifier en raison des faibles constantes d’association du ligand pour le Mn(II), un ion d5 ne présentant pas d’énergie de stabilisation du champ de ligand (LFSE), d’une grande labilité du ligand et d’un échange métallique rapide. La connaissance de la spéciation d’un complexe de Mn(II) ou d’un ion Mn(II) dans un environnement biologique est donc à la fois un réel défi et cruciale.
Au sein du Laboratoire des BioMolécules (LBM), le groupe multidisciplinaire « Métaux en biologie et homéostasie redox » développe des antioxydants catalytiques à base de métaux et bioinspirés, à savoir des complexes de manganèse(II) qui imitent les superoxyde dismutases (SODs). Ces métalloenzymes catalysent la dismutation du superoxyde, une espèce réactive de l’oxygène (ROS) dérivée de O2, et constituent la première ligne de défense antioxydante cellulaire. Le mime SOD noté Mn1 (voir figure) présente de bonnes activités intrinsèques anti-superoxyde, antioxydantes et anti-inflammatoires dans les cellules épithéliales intestinales HT29-MD2 activées par le lipopolysaccharide (LPS). Ce modèle cellulaire est utilisé pour évaluer les SOD mimétiques, et développé en collaboration avec l’équipe Microbiote, Intestin et Inflammation du Centre de Recherche de l’hôpital Saint-Antoine (UMRS 938, SU/INSERM).
Les méthodes de quantification utilisées précédemment (RPE, ICP-MS, XRF) par l’équipe pour caractériser des mimes de SODs étaient axées sur le contenu cellulaire en ions métalliques et sa comparaison avec le contenu endogène, en supposant que l’excès de Mn était dû au Mn1 internalisé.
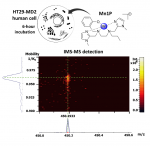
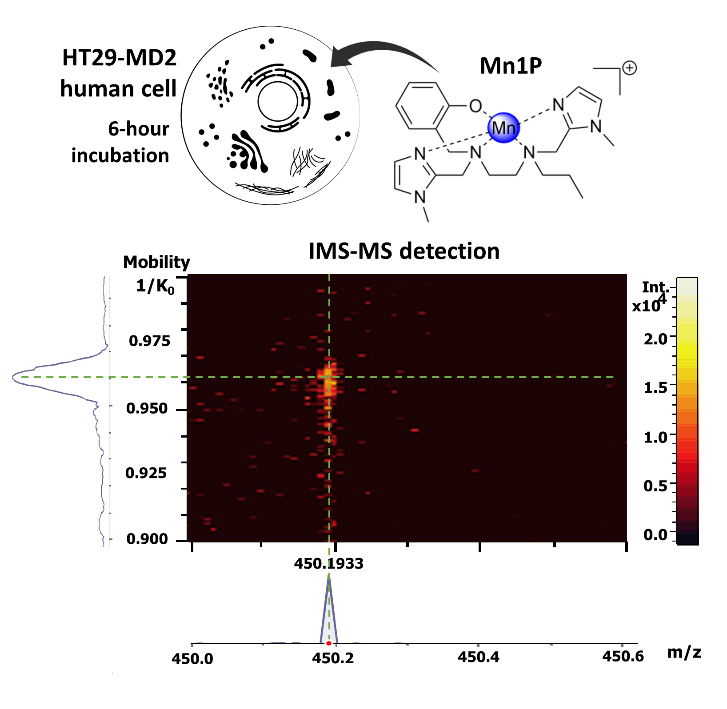
L’étude publiée récemment dans Angewandte Chemie (International Edition) comme une « very important publication » vise à élucider le devenir de Mn1 et de son analogue propylé noté Mn1P (qui a montré une activité similaire à Mn1) dans les lysats cellulaires HT29-MD2 en utilisant des méthodes de détection directe des entités moléculaires Mn1 et Mn1P basées sur la spectrométrie de masse (MS). L’équipe de chimistes bio-inorganiciens du LBM s’est associée à l’Institut des Sciences Analytiques et de Physico-Chimie pour l’Environnement et les Matériaux à Pau et au laboratoire de spectrométrie de masse biologique et protéomique (SMBP, ESPCI-PSL).
En raison de la complexité des milieux biologiques au voisinage des masses des complexes étudiés, il est apparu nécessaire de coupler la MS avec une méthode de séparation. Les techniques classiques de LC-MS (liquid chromatography coupled to mass spectrometry) n’ont pas donné satisfaction : les auteurs ont montré que des échanges métalliques se produisent dans le système analytique. Ils ont alors envisagé un couplage avec la spectrométrie de mobilité ionique (IMS). Cette méthode, développée pour la première fois par Cohen et Karasek en 1970 pour détecter des traces de molécules organiques en phase gazeuse, implique le mouvement des ions contre un flux gazeux sous un champ électrique. Dans le cas du présent travail, utilisant un timsTOF, les complexes métalliques sont poussés par le flux de gaz et retenus par un champ électrique. La vitesse des ions est proportionnelle à leur constante de mobilité réduite K (cm-1V-1s-1), qui est associée à leur taille et plus spécifiquement à leur section transversale de collision (CCS, en Å2).
Les auteurs montrent ici que la mobilité ionique est assez puissante pour distinguer des complexes du même ligand lié à différents cations métalliques divalents qui ont pourtant des rayons ioniques similaires (typiquement Mn(II), Fe(II), Co(II), Cu(II), Zn(II)).
Ils l’appliquent, en couplage avec la MS, pour démontrer la présence intracellulaire des complexes Mn1 et Mn1P dans les lysats de cellules incubées avec les complexes. Même s’ils mettent en évidence expérimentalement des échanges avec les métaux endogènes, les complexes ont été détectés. Dans le cas de Mn1P, grâce à une courbe d’étalonnage impliquant un analogue lourd (Co-13C6-ligand), ils ont pu estimer la teneur en Mn1P dans les cellules à 0,013 ± 0,006 fmol/cellule.
Voir aussi : https://www.inc.cnrs.fr/fr/cnrsinfo/des-complexes-bio-inspires-dans-le-vent
Source :
Deciphering the Metal Speciation in Low-Molecular-Weight Complexes by IMS-MS: Application to the Detection of Manganese Superoxide Dismutase Mimics in Cell Lysates
Martha Zoumpoulaki, Gabrielle Schanne, Nicolas Delsuc, Hugues Preud’homme, Elodie Quévrain, Nicolas Eskenazi, Géraldine Gazzah, Regis Guillot, Philippe Seksik, Joelle Vinh, Ryszard Lobinski, Clotilde Policar*
[*] Prof. C. Policar, Laboratoire des biomolécules (LBM), Département de chimie, École normale supérieure, PSL University, Sorbonne Université, CNRS, 75005 Paris, France, email : clotilde.policar@ens.psl.eu
Dr. M. Zoumpoulaki, G. Schanne, Dr. N. Delsuc, Laboratoire des biomolécules (LBM), Département de chimie, École normale supérieure, PSL University, Sorbonne Université, CNRS, 75005 Paris, France
Dr. M. Zoumpoulaki, Dr. N. Eskenazi, Dr. J. Vinh, SMBP ESPCI Paris, PSL University, UMR 8249 CNRS
Dr. M. Zoumpoulaki, G. Schanne, Prof./MD P. Seksik, Centre de Recherche de Saint-Antoine, Sorbonne University, INSERM, 75012 Paris (France)
Dr. H. Preud’homme, Prof. R. Lobinski, IPREM-UMR5254, E2S UPPA, CNRS, Technopôle Helioparc, 64053 Pau Cedex 9 (France)
Dr. R. Guillot, Université Paris-Saclay, CNRS, 91405 Orsay (France)
